Innym kompleksem nieredukowalnie złożonym jest spliceosom. Najpierw wyjaśnię czym jest spliceosom i jaką spełnia funkcję w żywych komórkach. Jest to proces znany i opisany w wielu artykułach naukowych i prawie wszystkich podręcznikach do podstaw biologii i biologii molekularnej. Nie będę więc wyważał otwartych drzwi i opis działania tego kompleksu zaczerpnę z internetu;
http://pl.wikipedia.org/wiki/Splicing
„Splicing, składanie genu, wycinanie intronów – usunięcieintronów (sekwencji niekodujących) i połączenie eksonów (sekwencji kodujących) z prekursorowego mRNA organizmów eukariotycznych. Proces ten zachodzi podczas obróbki posttranskrypcyjnej po to, by dojrzały mRNA, przygotowany do translacji, kodował ciągły łańcuch polipeptydowy (od kodonu start do stop). Splicing katalizowany jest przez kompleks białek i RNA zwany spliceosomem.”
http://upload.wikimedia.org/wikipedia/c ... ram_pl.svg
Zanim opis przejdzie w fazę zaawansowaną, to najpierw wyjaśnię, co to jest matrycowy RNA oraz co to są introny i egzony. Otóż przed powstaniem białka gen zapisany w DNA w procesie transkrypcji jest przepisywany na matrycowy RNA. Dopiero później na bazie tego matrycowego RNA powstaje białko na złożonym kompleksie biologicznym, który nazywamy rybosomem. Na tej animacji można zobaczyć jak po kolei zachodzą te procesy:
http://www.youtube.com/watch?v=GkdRdik73kU
Kiedy gen zostanie przepisany na matrycowy RNA wówczas matrycowe RNA składa się na przemian z odcinków kodujących [egzonów] oraz z odcinków niekodujących [intronów]. Specjalna maszyneria biologiczna, złożona z kilku białek ma zadanie wyciąć niepotrzebne fragmenty matrycowego RNA [introny], które następnie ulegają degradacji w komórce, i połączyć te potrzebne [egzony].
Matrycowe RNA przed wycięciem intronów i połączeniu egzonów nazywamy pre-mRNA ;
Wycinanie intronów przez spliceosom
http://upload.wikimedia.org/wikipedia/c ... ram_pl.svg
„Intron, by podlegał poprawnemu wycięciu, musi posiadać sygnały: sekwencję GU na końcu 5' i sekwencję AG na końcu 3' (dla spliceosomu klasycznego, odpowiedzialnego za przeważającą większość reakcji wycinania intronów, natomiast dla spliceosomu alternatywnego odpowiednio AU i AC) oraz tzw. miejsce rozgałęzienia, w którym znajduje się nukleotyd adeninowy (A). Podczas reakcji splicingu sekwencje sygnałowe są rozpoznawane przez wchodzące w skład spliceosomu małe jądrowe RNA (snRNA), które tworzą komplementarne połączenia RNA-RNA z obszarami terminalnymi i miejscem rozgałęzienia intronu. Splicing dokonywany przez spliceosom zaczynają się od ataku grupy 2'OH nukleotydu adeninowego z miejsca rozgałęzienia na pierwszy nukleotyd intronu (koniec 5'), co powoduje powstanie pętli. Następnie grupa 3'-OH uwolnionego eksonu atakuje ostatni nukleotyd intronu na końcu 3', dzięki czemu eksony się łączą i uwalniany jest intron w formie lassa.”
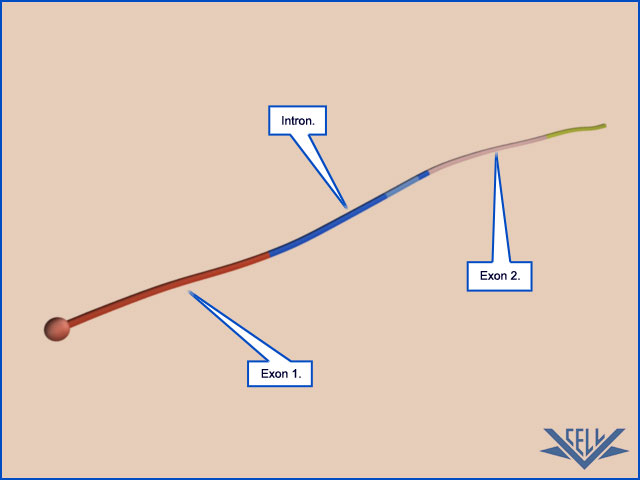
Pre-mRNA zawierające introny i egzony [oznaczone niebieskim kolorem]
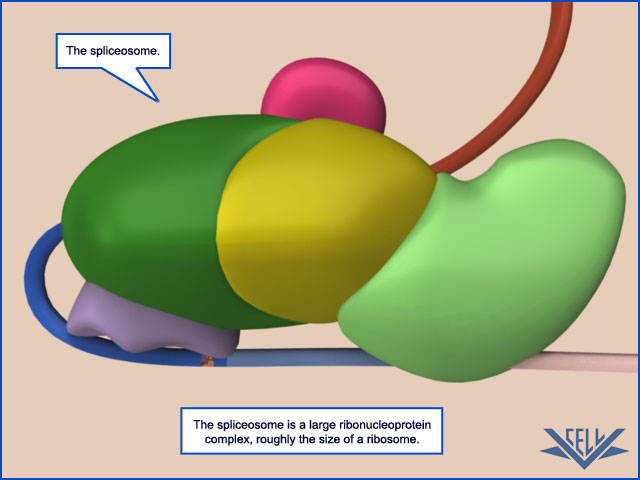
Spliceosom przyłącza się do pre-mRNA i formuje pętlę
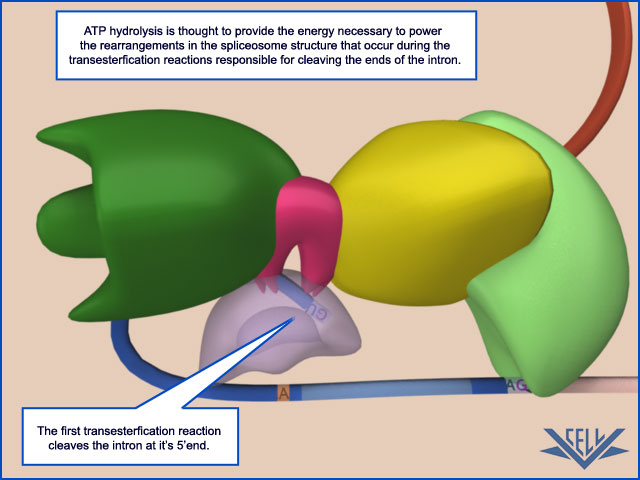
Następuje przecięcie intronu na styku inron-egzon końcu 5' pre-mRNA
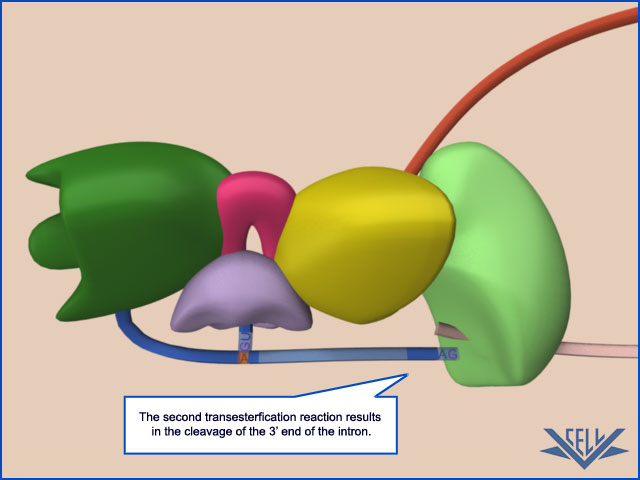
Na końcu 3' następuje również oddzielenie intronu od egzonu
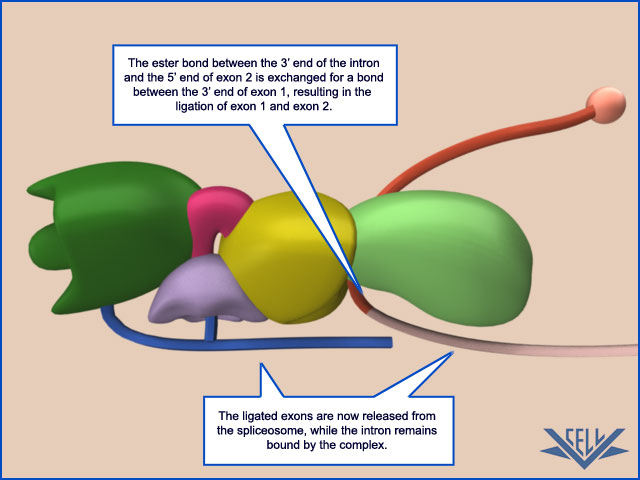
Po wycięciu intronów z pre-mRNA i połączeniu egzonów powstaje gotowe do translakcji matrycowe RNA na białko;
http://www.youtube.com/watch?v=Jml8CFBWcDs
Tutaj spliceosom w akcji można zobaczyć na filmie, który jest animacją obrazującą ten proces:
http://www.youtube.com/watch?v=FVuAwBGw_pQ
Na koniec dodam, że istnieją introny, które same potrafią się wycinać, gdyż ich sekwencje tworzą odpowiednie RNA enzymy, to znaczy rybozymy. Mimo, że niektore introny wycinające się bez udziału spliceosomu uważa się za starsze ewolucyjnie niż splicing z udziałem kompleksu białkowego , to taki proces nie mógłby być prekursorem ewolucyjnym splicingu z udziałem białek. Splicing z udziałem rybozymów sprawdza się w niewielkiej ilości prostych procesów. Jeżeli chodzi o splicing z udziałem spliceosomu, to jest to proces nieredukowalnie złożony, ponieważ żadne z białek biorących w nim udział nie może spełniać osobno swojej roli pod względem pełnionej funkcji (ani żadnej innej funkcji), z kolei, gdy się usunie jedno białek z kompleksu tworzącego spliceosom, również przestanie on spełniać swoją funkcję (i nie będzie pełnił żadnej innej funkcji w komórce). Wniosek z tego taki, że spliceosom nie mógł ewoluować stopniowo od prostszych prekursorów, poprzez dodawanie do układu kolejnych elementów.
Zaznaczam, że Michael Behe, ani żaden inny biolog i teoretyk inteligentnego projektu w przyrodzie, z tego, co mi wiadomo, nie używał przykładu spliceosomu, jako kompleksu nieredukowalnie złożonego. Jak zdecydowałem się używać tego przykładu na podstawie gruntownej analizy poświęconej temu zjawisku, jak i na podstawie wielu dyskusji z biologami / ewolucjonistami. Nigdy nie trafiłem na szczegółowe wyjaśnienia, jak mógł ewoluować spliceosom od prostszych prekursorów, więc zdecydowałem się zacząć używać tego przykładu, ponieważ uważam, że jest to pozycja sprawdzona pod względem argumentacji na rzecz nieredukowalnej złożoności kompleksów biologicznych.
